Ilościowa reakcja łańcuchowa polimerazy w czasie rzeczywistym (ang. real time quantitative polymerase chain reaction, qPCR) to metoda analityczna szeroko stosowana w diagnostyce oraz badaniach naukowych. W metodzie tej poziom ekspresji badanych genów jest wyznaczany w odniesieniu do genu lub genów referencyjnych. Zakłada się, iż poziom tych genów jest stały niezależnie od czynników środowiskowych, jakim podlega komórka, lub od jej stanu fizjologicznego Dobór właściwego genu/genów kontrolnych jest bardzo ważnym etapem. Dokonany jedynie na podstawie literatury, może skutkować błędnym wyznaczeniem poziomu ekspresji genów badanych, a co za tym idzie – błędną interpretacją wyników.
W opracowanym narzędziu stosuje się rozbicie grupy analizowanych próbek (model eksperymentalny) na szereg podmodeli składających się z mniejszej liczby próbek (minimalnie z dwóch), a następnie wytypowanie w każdym z tych modeli najlepszego genu referencyjnego z grupy analizowanych w danym eksperymencie potencjalnych genów referencyjnych, z wykorzystaniem znanego algorytmu Norm- Finder. Wytypowany w ten sposób gen lub para genów będących najlepszą referencją w danym modelu stanowi następnie podstawę do obliczenia ekspresji względnej genów badanych w poszczególnych modelach i przeprowadzenia walidacji spójności otrzymanych wyników.
W ramach każdego z modeli prowadzone są analizy statystyczne bazujące na odpowiednio dobranych testach, pokazujące istotności statystyczne różnic w ekspresji genu badanego w obrębie danego modelu. Kolejnym etapem jest porównanie spójności otrzymanych wyników/wartości i ich statystycznej istotności dla danego genu badanego w obrębie wszystkich badanych modeli. Program wyznacza tzw. coherence score (współczynnik spójności) opisujący poziom spójności otrzymanych wyników. Osiągnięcie niezadowalającej wartości tego współczynnika – poniżej akceptowalnej przez badacza – prowadzi do kolejnego etapu analizy, tj. ponownego wyboru genów referencyjnych we wszystkich badanych modelach, ale odbywa się on po uprzednim usunięciu z listy potencjalnych genów referencyjnych tego, który na poprzednim etapie uzyskał najsłabszy poziom stabilności (czyli np. z grupy wyjściowych n genów referencyjnych usuwany jest ten o najsłabszym wyniku stabilności i następnie właściwy gen referencyjny jest wybierany spośród pozostałych n-1 potencjalnych genów referencyjnych).
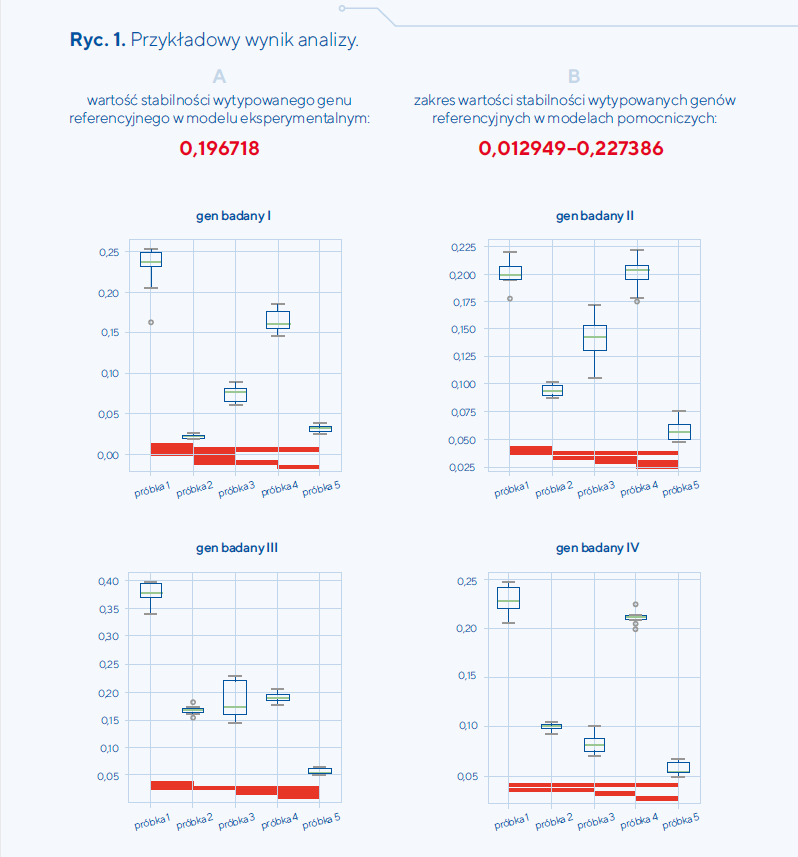
Nowe rozwiązanie łączy w sobie funkcjonalności programów wyznaczających najlepsze geny referencyjne oraz programów typu arkusze kalkulacyjne, które pozwalają na analizę poziomu ekspresji genu badanego, analizę statystyczną i interpretację graficzną wyników. Jego istotnymi elementami są zaimplementowanie określonego schematu analizy danych RT-qPCR (usuwanie genów o najsłabszej stabilności z następującą po tym analizą danych surowych) oraz wprowadzenie wartości współczynnika spójności określającego rzetelność/wiarygodność przeprowadzonej analizy. Usprawnieniem całości procesu jest też automatyczne generowanie tabel oraz wykresów pudełkowych prezentujących otrzymane wyniki analizy, włącznie z zaznaczonymi na wykresie istotnościami statystycznymi między badanymi elementami danego modelu (ryc. 1). Program umożliwia jednoczesną i niezależną analizę wielu genów badanych.
Opracowane narzędzie informatyczne umożliwia:
1) selekcję referencji, o ulepszonej wartości stabilności, z wyjściowej puli potencjalnych genów referencyjnych;
2) określenie rzeczywistego poziomu ekspresji genów badanych;
3) prawidłową interpretację biologiczną wyników na podstawie kompleksowej analizy danych surowych otrzymanych w reakcji RT-qPCR dla wieloelementowych modeli eksperymentalnych (duże grupy badawcze złożone z linii komórkowych, próbek pobranych od pacjentów lub zwierząt).
Poniżej (po kliknięciu) ulotka promująca wynalazek.
Program został niniejszym udostępniony nieodpłatnie na zasadach tzw. trzyklauzulowej licencji BSD.
Copyright 2021 Uniwersytet Jagielloński, Dr hab. Dorota Hoja-Łukowicz, prof. UJ, Dr Marcelina Janik.
1 Redystrybucje kodu źródłowego muszą zawierać powyższą informację o prawach autorskich, niniejszą listę warunków i poniższe zastrzeżenie.
2. Redystrybucje w formie binarnej muszą zawierać powyższą informację o prawach autorskich, tę listę warunków i poniższe wyłączenie odpowiedzialności w dokumentacji i/lub innych materiałach dostarczonych wraz z dystrybucją.
3. Ani nazwa Instytutu, ani imiona jego współpracowników nie mogą zostać użyte do wspierania i promocji produktów opartych na tym oprogramowaniu bez wcześniejszego uzyskania konkretnej pisemnej zgody.
GenExpA.zip

informacja / kontakt broker Uniwersytetu Jagiellońskiego